国際ゲノム解析により関節リウマチの遺伝的背景を解明-個人のゲノム情報を活用した発症予測の社会実装に貢献-
- ヘッドライン
- 記者発表
理化学研究所
大阪大学
東京大学
京都大学
産業医科大学
東京医科歯科大学
東京女子医科大学
ハーバード大学
発表概要
理化学研究所(理研)生命医科学研究センターヒト免疫遺伝研究チームの石垣和慶チームリーダー、大阪大学大学院医学系研究科の坂上沙央里助教(ハーバード大学医学部博士研究員)、理研生命医科学研究センターゲノム解析応用研究チームの寺尾知可史チームリーダー、東京大学大学院新領域創成科学研究科の松田浩一教授、京都大学大学院医学研究科の松田文彦教授、産業医科大学医学部の田中良哉教授、大阪大学大学院医学系研究科の熊ノ郷淳教授、東京医科歯科大学難治疾患研究所の高地雄太教授、東京女子医科大学医学部の猪狩勝則特任教授、理研生命医科学研究センター自己免疫疾患研究チームの山本一彦チームリーダーらが参加し、理研生命医科学研究センターシステム遺伝学チームの岡田随象チームリーダー(大阪大学大学院医学系研究科教授)とハーバード大学のショウモウ・レイチャウドリ教授らが統括する国際共同研究グループは、関節リウマチの発症に関わる34個の遺伝的変異を新たに同定しました。
本研究成果は、関節リウマチの新しい治療標的の同定や、個人の遺伝情報に基づく個別化医療の発展に貢献すると期待できます。
今回、国際共同研究グループは、日本人集団を含む五つの人種集団から構成される約28万人のゲノム解析を行い、関節リウマチの発症に関わる34個の遺伝的変異を新たに同定しました。さまざまなデータベースとの統合解析を行うことで、関節リウマチの発症に関与する分子メカニズムを多数解明しました。
さらに、個々人の遺伝子情報から関節リウマチの発症を予測するリスクスコアを構築し、東アジア人集団における予測精度が欧米人集団と同程度であることを確認しました。
本研究は、科学雑誌『Nature Genetics』オンライン版(2022年11月4日付)に掲載されました。
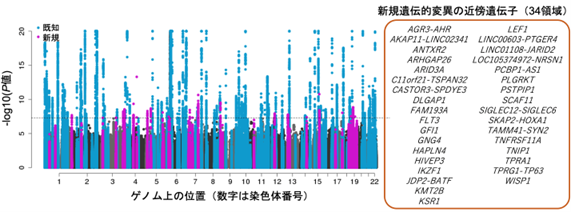
関節リウマチの発症に関わる新しい遺伝子の候補
発表内容
研究の背景
関節リウマチは、免疫システムが関節組織を破壊して機能が損なわれる原因不明の疾患です。
関節リウマチの発症には、多くの遺伝的変異(リスク変異)が関与することが知られています。そのため、ゲノムワイド関連解析(GWAS)[1]によってリスク変異を同定する研究が欧米を中心に行われてきました。しかし、大半のリスク変異の影響は非常に小さいため、従来のGWAS研究では一部のリスク変異しか同定できませんでした。また、単一の人種集団を対象とするGWASでは、ゲノム上の近傍に位置する遺伝的変異間の相関関係のため、リスク変異をピンポイントで同定することは一般的に困難であり、その局在も不明瞭でした。
一方、多くの患者・健常者のサンプルを多人種集団のコホート(集団)から集め、大規模にGWASを実施することで、変異間の相関関係の影響が緩和され、より多くのリスク変異を高い精度で同定できることが知られています。
近年、欧米人集団を対象に大規模なGWASが多数実施され、その解析結果は、疾患発症の予測などの目的で、臨床現場で実用性が示されつつあります。一方、遺伝的変異の分布には人種差があるため、欧米人集団での解析結果を日本人集団に応用できる範囲は限定的です。そのため、日本において関節リウマチのゲノム医療を実現するためには、多くの東アジア人集団をGWASの対象に含めることが必須だと考えられています。
このような背景から本研究では、日本人集団を含む東アジア人集団など五つの人種集団を対象とした関節リウマチのGWASを過去最大規模で実施しました。
研究手法と成果
本研究には、欧米人集団、東アジア人集団、アフリカ人集団、南アジア人集団、アラビア人集団から構成される37コホートが参加し、35,871人の関節リウマチ患者と240,149人の健常者、合計276,020人を対象にGWASを実施しました(図1)。これは関節リウマチを対象としたものでは過去最大規模のものです。多くのGWASが欧米人集団中心に行われているのに対して、今回は患者の約3分の1(11,025人)が東アジア人集団であり、その大半は日本で収集されたサンプルでした。
このGWASによって、合計124個のリスク変異が同定され、そのうち34個が新規の発見でした。
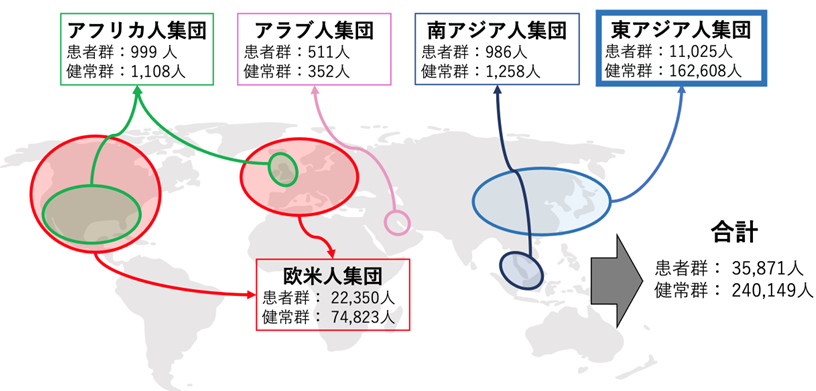
図1 本研究で解析した人種集団ごとのサンプル数
今回のGWASに用いられた各人種集団のサンプル数を患者群・健常群に分けて示した。サンプルが収集された施設の地理情報も示している。
さらに国際共同研究グループは、検出されたリスク変異の局在を近似ベイズ因子[2]を用いて詳細にマッピングしました。すると予想通り、単一人種集団でのGWASと比較して、多人種集団でのGWASでは、より精度の高いマッピング結果が得られました(図2a)。
本研究全体で、事後確率[3]が50%を超える35個のリスク変異を同定することに成功しました。
最も高い精度でマッピングされたリスク変異はLEF1遺伝子座に同定されました。LEF1遺伝子変異はCD4陽性T細胞[4]の遺伝子発現制御領域[5]にあることから、CD4陽性T細胞の遺伝子発現制御異常が発症リスクに関わることが示唆されました(図2b)。
リスク変異の局在を高精度にマッピングすることは、変異の機能解析実験において重要な情報となることから、本研究成果は関節リウマチの病態解明の加速につながると考えられます。
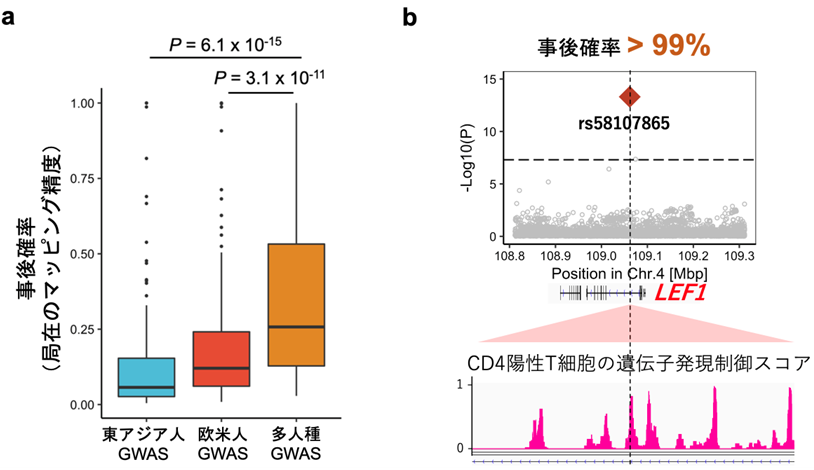
図2 リスク変異の局在のマッピング
a) 局在のマッピング精度である事後確率をGWAS条件ごとに比較した。122個のリスク変異の事後確率の分布を示している。東アジア人集団だけを対象としたGWAS、欧米人集団だけを対象としたGWAS、5人種集団全てを含めたGWASの結果を用いた。各条件間の差の有意性をWilcoxon検定(1対のサンプルの差を評価するノンパラメトリック検定法の一つ)で評価し、そのP値を示した。P値は統計検定において、帰無仮説の下で検定統計量が観測された値となる確率のことで、P値が小さいほど、観測された現象がまれである(ここでは、2つの条件下でマッピング精度が異なる可能性が高い)ことを意味する。
b) 99%以上の事後確率が得られたリスク変異の一例。rs58107865はLEF1遺伝子領域に存在し、CD4陽性T細胞の遺伝子発現の制御活性が高い領域にある。
リスク変異が引き起こす分子機序の解明を目指したところ、PADI4遺伝子座のリスク変異において興味深い知見が得られました。関節リウマチの最も特徴的な免疫異常が、シトルリン化[6]されたタンパク質に対する自己抗体[7]の存在です。PADI4遺伝子はシトルリン化を担う酵素をコードしている非常に重要な遺伝子です。英国のプロジェクトBLUEPRINT[8]のデータベースとの統合解析によって、PADI4遺伝子変異は好中球[9]でPADI4遺伝子のスプライシング[10]に関与する可能性が示されました。しかし、PADI4スプライシングアイソフォーム[10]の全体像は不明であり、詳細な分子機序を解明することができませんでした。そこで、ロングリードシーケンス[11]の技術を用いて、PADI4遺伝子の転写産物の全長配列を確認し、酵素活性に重要な部分を欠く新しい非機能性スプライスアイソフォームを特定しました(図3a)。
次に、105人の日本人集団の健常者からのRNAシーケンスデータ[12]を使用して、PADI4アイソフォームをそれぞれ定量評価しました。すると、リスク対立遺伝子[13]は、非機能性アイソフォームの発現量の減少、機能性アイソフォームの発現量の増加、全アイソフォームの合計発現量の減少と関連していることが分かりました(図3b)。
つまり、アイソフォームごとの詳細な定量評価により、リスク対立遺伝子はPADI4遺伝子の機能を亢進させることが示唆されました。この結果は、PADI4遺伝子の新しい遺伝的メカニズムを解明し、ロングリードシークエンスを使用して、リスク遺伝子座でスプライスアイソフォームを徹底的に調査することの重要性を示しています。
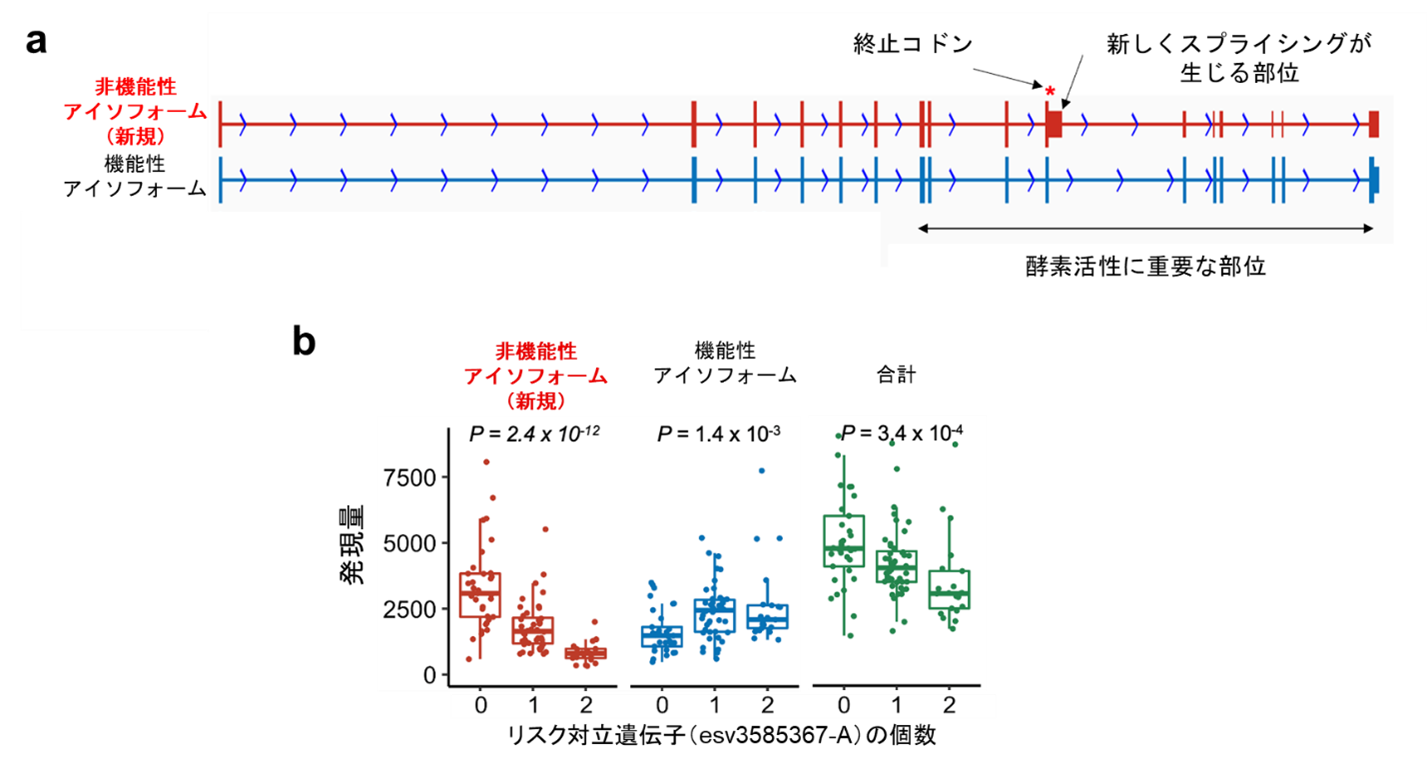
図3 PADI4 遺伝子で同定された複数種類のアイソフォーム
a) 新たに同定された非機能性アイソフォーム(赤)と機能性アイソフォーム(青)の構造。
b) リスク対立遺伝子(esv3585367-A)の個数と、新たに同定された非機能性アイソフォームの発現量(赤)、機能性アイソフォームの発現量(青)、全アイソフォームの合計発現量(緑)との関連を線形回帰解析で評価した。発現量は好中球を多く含む全血のRNAシーケンスによって求めた。リスク対立遺伝子は、非機能性アイソフォームの発現量の減少、機能性アイソフォームの発現量の増加、全アイソフォームの合計発現量の減少と関連している。
最後に、今回の研究成果によって各人種集団の発症リスクがどの程度予測可能かを検証するため、ポリジェニック・リスク・スコア(PRS)[14]を計算しました。PRSはGWASの結果と個人の遺伝子変異情報を基に発症リスクを推定する指標であり、他の疾患では臨床現場での実用性が示されつつあります。過去、多くの研究において東アジア人集団のPRS予測精度は欧米人集団よりも低くなることが多かったのですが、今回、東アジア人集団と欧米人集団とで同程度のPRS予測精度が観察されました(図4)。これは、バイオバンク・プロジェクトなど日本人集団を中心とした東アジア人集団のサンプルを多く収集することに成功したからだと考えられます。
なお、このPRS計算では、発症リスクに強い影響を与えるHLA遺伝子[15]変異の情報は用いていません。今後、HLA遺伝子変異の情報を加えることで、予測精度はさらに高まると考えられます。
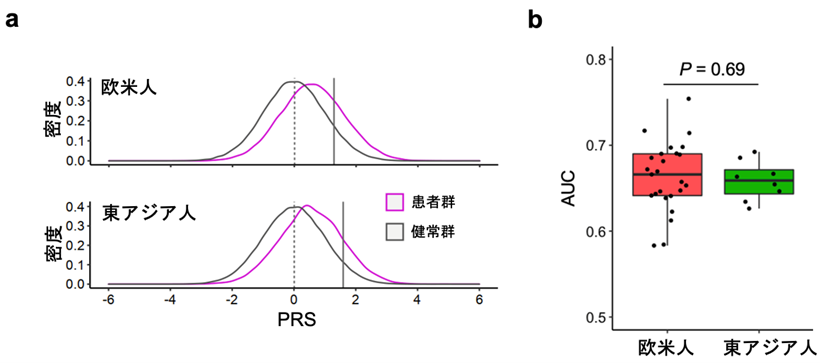
図4 ポリジェニック・リスク・スコア(PRS)の欧米人集団と東アジア人集団との比較
a) 欧米人集団と東アジア人集団のPRSの分布を患者群・健常群に分けて示す。
b) PRSの予測精度能を欧米人集団・東アジア人集団の各コホートで、AUC(Area Under the Curve)を用いて評価・比較した。AUCは分類法における精度の指標として用いられる。0.5から1の値をとり、1が最も精度が高いことを意味する。Wilcoxon検定で有意な差がないことが確認された。
今後の期待
本研究結果は関節リウマチの発症に関わる生物学的機序を解明し、ゲノム創薬に貢献すると考えられます。
本研究結果は公共データベースで全て公開されるため、今後世界的に活用され多方面の研究活動を促進することが期待できます(GWAS Catalog: https://www.ebi.ac.uk/gwas/)。
さらに、PRSの発症予測精度が東アジア人集団と欧米人集団とで同程度であった結果から、日本におけるゲノム医療の実現に向けた議論を加速させることも期待できます。
研究支援
本研究は、日本医療研究開発機構(AMED)免疫アレルギー疾患実用化研究事業「疾患ゲノム情報を活用した自己免疫疾患における核酸ゲノム創薬の推進(研究代表者:岡田随象)」「免疫オミクス情報の横断的統合による関節リウマチのゲノム個別化医療の実現(研究代表者:岡田随象)」、同ゲノム医療実現推進プラットフォーム事業(先端ゲノム研究開発:GRIFIN)「遺伝統計学に基づく日本人集団のゲノム個別化医療の実装(研究代表者:岡田随象)」、大阪大学大学院医学系研究科バイオインフォマティクスイニシアティブによる助成を受けて行われました。
論文情報
<タイトル>
Multi-ancestry genome-wide association analyses identify novel genetic mechanisms in rheumatoid arthritis
<著者名>
Kazuyoshi Ishigaki, Saori Sakaue, Chikashi Terao, Yang Luo, Kyuto Sonehara, Kensuke Yamaguchi, Tiffany Amariuta, Chun Lai Too, Vincent A. Laufer, Ian C. Scott, Sebastien Viatte, Meiko Takahashi, Koichiro Ohmura, Akira Murasawa, Motomu Hashimoto, Hiromu Ito, Mohammed Hammoudeh, Samar Al Emadi, Basel K. Masri, Hussein Halabi, Humeira Badsha, Imad W. Uthman, Xin Wu, Li Lin, Ting Li, Darren Plant, Anne Barton, Gisela Orozco, Suzanne M. M. Verstappen, John Bowes, Alexander J. MacGregor, Suguru Honda, Masaru Koido, Kohei Tomizuka, Yoichiro Kamatani, Hiroaki Tanaka, Eiichi Tanaka, Akari Suzuki, Yuichi Maeda, Kenichi Yamamoto, Satoru Miyawaki, Gang Xie, Jinyi
Zhang, Christopher I. Amos, Ed Keystone, Gertjan Wolbink, Irene van der Horst-Bruinsma, Jing Cui, Katherine P. Liao, Robert J. Carroll, Hye-Soon Lee, So-Young Bang, Katherine A. Siminovitch, Niek de Vries, Lars Alfredsson, Solbritt Rantapää-Dahlqvist, Elizabeth W. Karlson, Sang-Cheol Bae, Robert P. Kimberly, Jeffrey C. Edberg, Xavier Mariette, Tom Huizinga, Philippe Dieudé, Matthias Schneider, Martin Kerick, Joshua C. Denny, The Biobank Japan Project, Koichi Matsuda, Keitaro Matsuo, Tsuneyo Mimori, Fumihiko Matsuda, Keishi Fujio, Yoshiya Tanaka, Atsushi Kumanogoh, Matthew Traylor, Cathryn M. Lewis, Stephen Eyre, Huji Xu, Richa Saxena, Thurayya Arayssi, Yuta Kochi, Katsunori Ikari, Masayoshi Harigai, Peter K. Gregersen, Kazuhiko Yamamoto, S. Louis Bridges, Jr, Leonid Padyukov, Javier Martin, Lars Klareskog, Yukinori Okada and Soumya Raychaudhuri
<雑誌>
Nature Genetics
<DOI>
10.1038/s41588-022-01213-w
用語解説
[1] ゲノムワイド関連解析(GWAS)
疾患や身長・体重などの形質に影響があるゲノム上のマーカー(遺伝的変異)を網羅的に検索する手法。
2002年に、理研が世界に先駆けて報告を行っており、以降、さまざまな疾患や量的形質に関連するゲノムマーカーの同定に貢献している。GWASはGenome-Wide Association Studyの略。
[2] 近似ベイズ因子
ベイズ統計学において用いられる数値であり、実験結果が仮説をサポートする強さを数値化したもの。
[3] 事後確率
事前に想定される確率と得られた実験結果から推定される事後的な確率。
[4] CD4陽性T細胞
胸腺で分化し、細胞表面にCD4を発現するリンパ球。免疫システム全体の活動を調整する機能がある。
[5] 遺伝子発現制御領域
プロモーターやエンハンサーなどの転写因子などが結合して、遺伝子の発現量を調整するゲノム上の領域。
[6] シトルリン化
翻訳後修飾の一種。タンパク質中のアルギニンがシトルリンへ変換される。
[7] 自己抗体
抗体は免疫グロブリンとも呼ばれ、正常な状態では異物に特異的に結合し、その異物を生体内から除去する。
自己の細胞ないし組織に対して産生される抗体を自己抗体と呼ぶ。
[8] BLUEPRINT
遺伝的変異が遺伝子発現量などに与える影響を免疫細胞において網羅的に評価した英国のプロジェクト。
[9] 好中球
白血球の一種であり細胞内に顆粒を持つ。炎症部に遊走・集積し、細菌などの異物を貪食し生体を防御する。
[10] スプライシング、スプライシングアイソフォーム
一つの遺伝子に複数のエクソン(タンパク質をコードする部分)が存在する場合に、RNA分子においてエクソン間にある領域を除き、エクソン同士を直接つなげる作業をスプライシングと呼ぶ。
スプライシング後に形成されるRNAの配列パターンをスプライシングアイソフォームと呼ぶ。
[11] ロングリードシーケンス
次世代シーケンス手法の一種。通常は100塩基程度しかシーケンスできないが、ロングリードシーケンスでは10,000塩基程度までシーケンスすることができる。
[12] RNAシーケンスデータ
RNA分子の配列情報を次世代シーケンサーで解析し、遺伝子発現量などを評価したデータ。
[13] 対立遺伝子
遺伝的変異における個々の染色体上のDNA配列のこと。
[14] ポリジェニック・リスク・スコア(PRS)
個人の遺伝情報に基づき、ヒトゲノム配列全体に分布する多数の遺伝子変異を統合することで計算される、疾患の発症などの表現型を予測するリスクスコア。個別化医療の鍵として注目されている。
PRSはPolygenic Risk Scoreの略。
[15] HLA遺伝子
HLAは、免疫反応をつかさどる細胞である白血球の表面に発現する分子。生体内における自己と非自己の認識や外来性の病原菌に対する免疫反応を制御している。